Mit KI gegen Krebs: Die UBC-OCEAN Challenge
Programmiersprache: Python
Genutzte Software: Pytorch, Scikit-Learn, Py WSI, Timm, OpenCV
Keywords: Histopathology, Cancer Classification, Computer Vision, Convolutional Neural Networks, Recurrent Neural Network, Neuronales Netz, KI, SVM, Boosting, t-SNE
Inhaltsverzeichnis:
- Wie KI Krebs erkennt: Die UBC-OCEAN Challenge
- Warum wurde gerade das als Aufgabe gewählt?
- Doch was gibt es überhaupt für Typen und warum ist das wichtig?
- Erster Ansatz: Neuronales Netz
- Erkennung von Krebs auf Zellbasis: Der Patches Ansatz
- ResNet, ConvNet, EfficientNet… Was die großen Modelle können
- Histologie als Werkzeug in der Krebsforschung
- Krebszellen vs. Gesunde Zellen (Segmentierung)
- 2 Stage Model: Erst Detektieren, dann Klassifizieren
- Krebs KI als Multi Instance Learning: HGBC/RNN/t-SNE
Wie KI Krebs erkennt: Die UBC-OCEAN Challenge
Wir alle kennen die Fähigkeiten von KI-Algorithmen bei Bilddaten: Smartphones erkennen, was gerade fotografiert wird und passen die Einstellungen der Kamera an. Autos erkennen Fußgänger, die den Weg kreuzen und bremsen automatisch ab. Hautscreening Apps warnen vor bösartigen Hautveränderungen… Die Liste könnte noch lange so weitergehen.
Nun gibt es hier eine Unterscheidung, denn Fußgänger zu erkennen, das schafft jeder von uns. Bösartige Leberflecke auf der Haut können nur von einem Experten (Dermatologen) bewertet werden. Eine KI kann bei beiden Aufgaben schon zuverlässigere und schnellere Vorhersagen als Menschen treffen, daher bietet es sich an, sich das als Assistenzsystem zu Nutze zu machen.
Die UBC (University of British Columbia) hat die OCEAN Challenge auf Kaggle gestartet. Dabei steht OCEAN für den unglaublich sperrigen Ausdruck “Ovarian Cancer SubtypE ClAssification and Outlier DetectioN”. Oder einfacher formuliert: Es geht um die Erkennung von Eierstockkrebs und die Detektion von seltenen Varianten.
Warum wurde gerade das als Aufgabe gewählt?
Dafür gibt es zwei Gründe: Erstens ist eine Krebserkrankung an den Eierstöcken bei Frauen eine der schwerwiegendsten Krebsarten, d.h. eine frühe und sichere Diagnosestellung ist extrem wichtig.
Und zweitens ist eben diese Diagnosestellung absolut nicht einfach. Ein Leberfleck kann mit dem bloßen Auge angeschaut und bewertet werden. Bei Ovarialkarzinomen muss eine Biopsie des Gewebes durchgeführt und das dann histologisch untersucht werden. Es müssen also Bilder eines Mikroskops von Histopathologen/-innen angeschaut werden und diese Mediziner sollten außerdem spezialisiert auf den gynäkologischen Bereich sein. Allein durch die Beschreibung sollte schon deutlich werden, dass solche Spezialisten selten und damit die Diagnosestellung kompliziert ist.
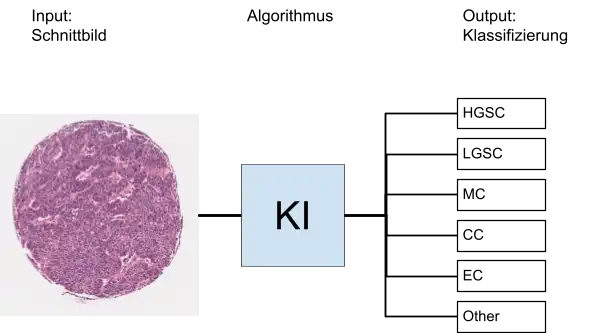
Damit wäre die Aufgabenstellung klar: Es soll eine KI erstellt werden, die aus histologischen Schnittbildern den Typ von Eierstockkrebs erkennt.
Doch was gibt es überhaupt für Typen und warum ist das wichtig?
Ein großer Bereich in der Erforschung von Krebstherapien widmet sich der Personalisierung, denn jeder Krebs ist unterschiedlich, auch wenn es der gleiche Typ ist. Daher sprechen manche Patienten sehr gut auf bestimmte Therapien an, während andere keinen Erfolg damit haben. Und so ist es auch bei Eierstockkrebs: Je weiter man den Typ eingrenzen kann, desto spezieller (und damit besser) kann man therapieren.
Grundlegend kann man hier 5 Typen unterscheiden:
- High Grade Serous Carcinoma (HGSC)
- Low Grade Serous Carcinoma (LGSC)
- Mucinous Carcinoma (MC)
- Endometrioid Carcinoma (EC)
- Clear Cell Carcinoma (CC)
Und dann gibt es noch mehrere seltene Tumorarten, die in dieser Challenge unter “Other” zusammengefasst werden. Hier ein sehr vereinfachter Überblick zu der Aufgabe der OCEAN Challenge:
Erster Ansatz: Neuronales Netz
Um eines gleich mal vorweg zu nehmen: Eine der großten Herausforderungen war nicht unbedingt der KI Algorithmus sondern das Preprocessing der Daten. Als Ausgangspunkt hatte man, wie oben schon erwähnt, Bilder von histologischen Schnitten unter dem Mikroskop. Diese Schnitte können mehrere cm groß sein und im besten Fall möchte man Zellen erkennen die in der Größenordnung von ein paar 𝛍m sind, d.h. man muss sehr tief zoomen und das funktioniert nur mit einer hohen Auflösung. Und ich spreche jetzt nicht von hohen Auflösungen, die man von Bildschirmen oder Kameras gewohnt ist. Hier gibt es Bilder mit bis zu 100.000 x 50.000 Pixel, was dann ca. 4GB Dateigröße bedeutet.
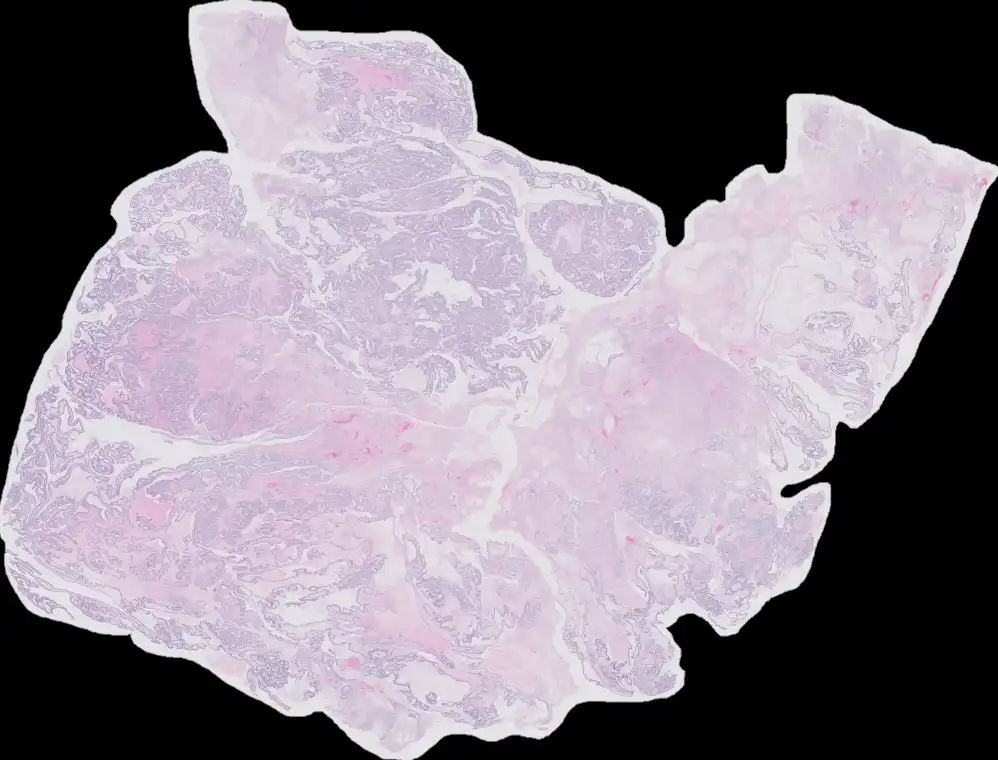
In den Trainingsdaten gab es aber auch Thumbnails, die um den Faktor 10 kleiner sind - auch nicht gerade handlich, aber für die ersten Versuche besser geeignet. Hier ein Beispiel dazu:
Insgesamt gibt es 538 Bilder und als erster Ansatz soll ein CNN (Convolutional Neural Network) darauf trainiert werden. Die Architektur sieht folgendermaßen aus:
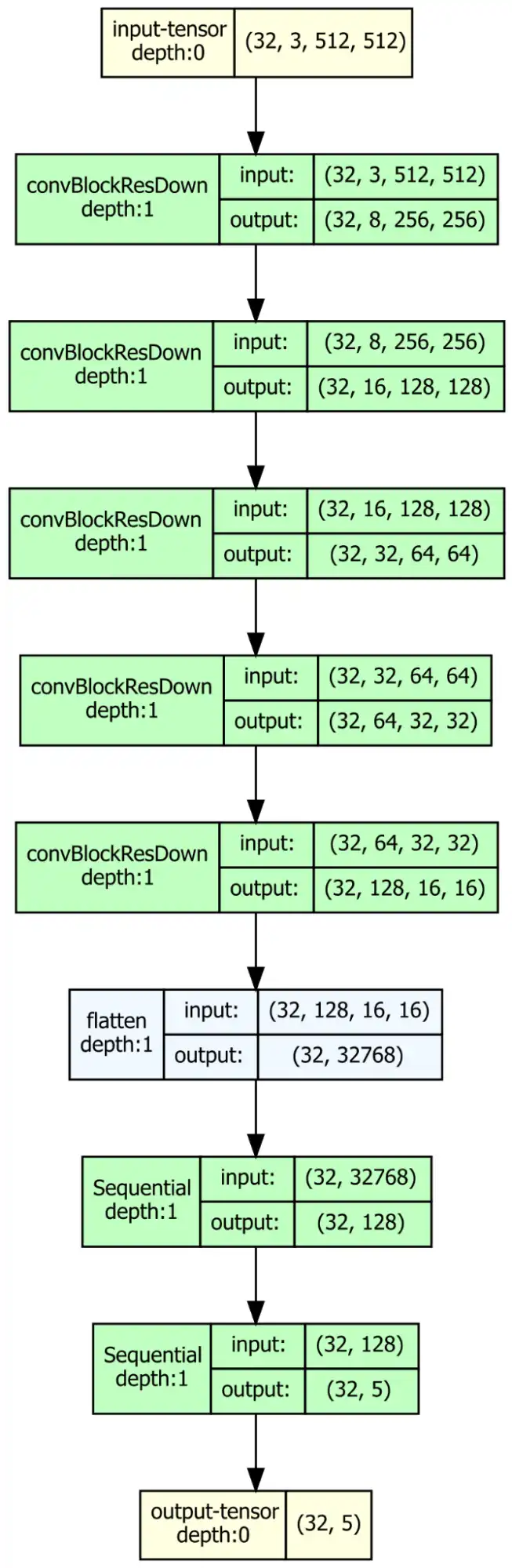
Wirklich gute Ergebnisse habe ich hier nicht erwartet, denn auf diesen makroskopischen Bildern erkennt man vielleicht, dass das Gewebe ungewöhnlich aussieht, aber für die Erkennung von Subtypen müssen einzelne Zellen betrachtet werden (dazu später mehr). Dementsprechend ergaben sich fürs Training diese Verläufe:
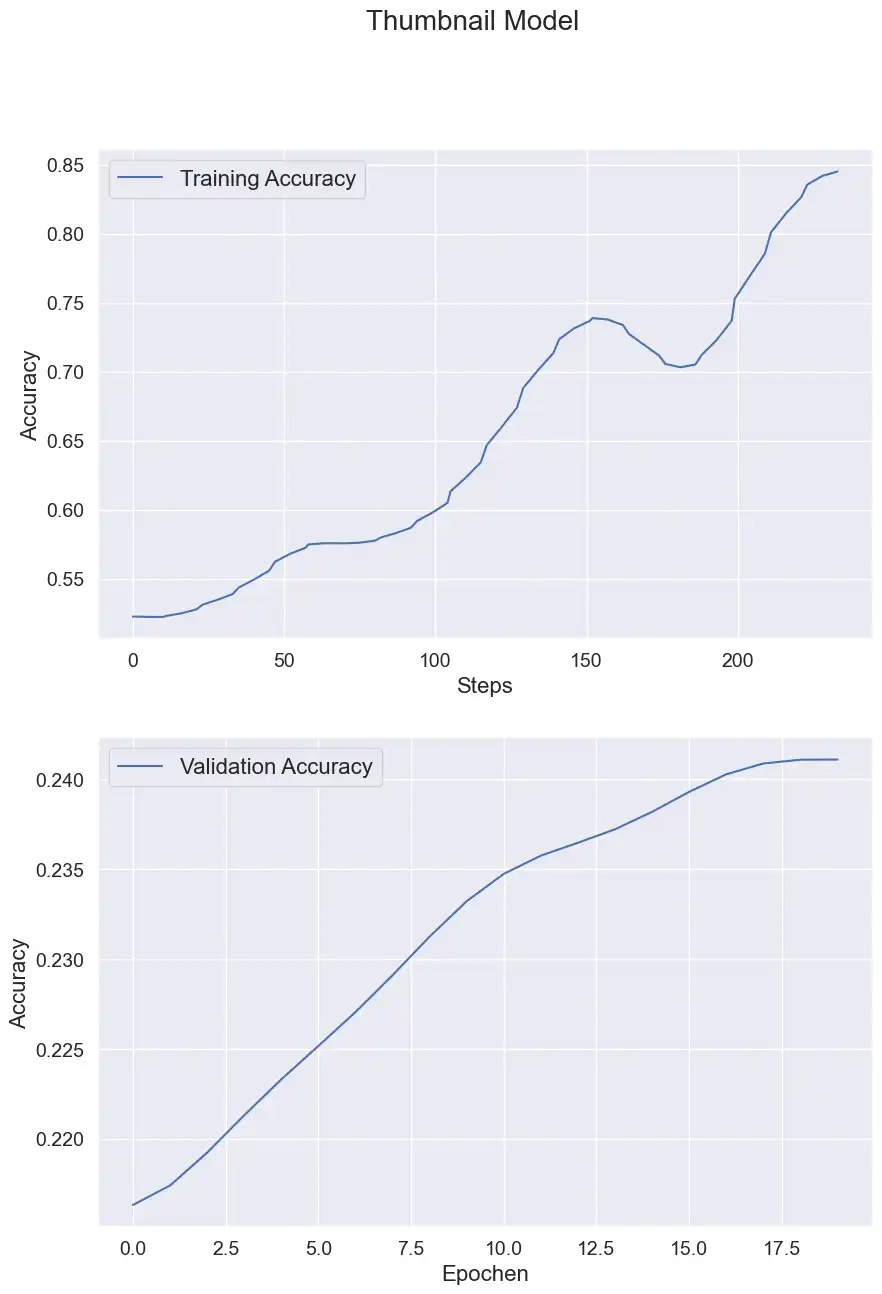
Hinweis: Bei allen Trainings wurden immer 70% der Daten für das Training und 30% der Daten für die Validierung aufgeteilt.
Als Metrik wurde in dieser Challenge immer Balanced Accuracy verwendet, d.h. für alle 5 (oder 6) Klassen wurde die Accuracy berechnet und davon dann der Durchschnitt. Damit ist es auch bei nicht gleichverteilten Klassen noch aussagekräftig.
Und wie erwartet schafft es das Thumbnail Modell gerade mal auf ca. 24%, obwohl es im Training deutlich besser performed hat. Hier ist anzunehmen, dass das Modell die Trainingsdaten overfitted hat, also Eigenschaften der Bilder gelernt hat, die nicht wirklich was mit der Aufgabe zu tun haben. Also z.B. die Form der Schnitte.
Es muss ein neuer Ansatz her - die relevanten Informationen stecken in den Details. Und genau diese Details muss man aus den riesigen Schnittbildern rausholen.
Erkennung von Krebs auf Zellbasis: Der Patches Ansatz
Die Idee ist einfach: Die riesigen Bilder werden in viele kleine quadratische Patches zerschnitten. In diesen “Patches” erkennt man sehr viel mehr Details auf Zellbasis, die das neuronale Netz lernen kann.
Die Patchgröße habe ich dafür auf 512px festgelegt und damit könnte man aus den größten Bildern jeweils 19.000 Patches extrahieren. Das ist eine riesige Datenmenge und der Trainingsprozess würde wahrscheinlich Wochen dauern, daher habe ich die Extraktion auf ein 7 x 7 Gitter beschränkt.
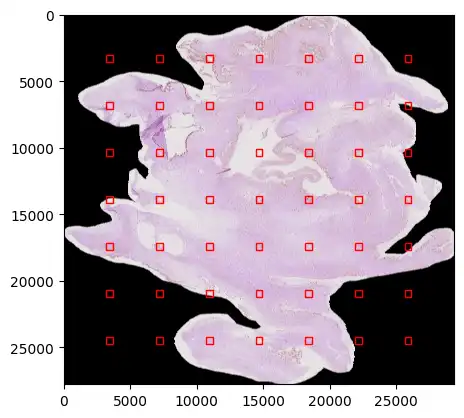
Wie im Bild oben zu sehen, wurden von jedem Bild 7 x 7 Patches (Größe 512px) in gleichmäßigem Abstand extrahiert.
Mit diesem Vorgehen gibt es auch einzelne Bilder außerhalb des Gewebes, welche dann komplett schwarz sind. Dafür habe ich beim Ladevorgang einen Filter eingebaut, der das Histogramm jedes Patches überprüft. Wenn der Großteil der Pixel schwarz ist, wird es übersprungen.
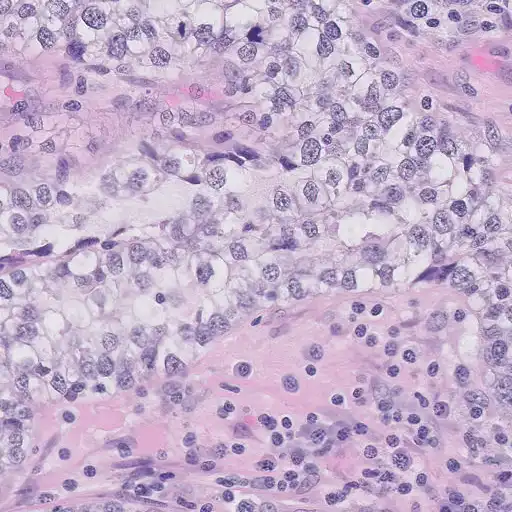
In diesem Beispiel-Patch erkennt man genau die Strukturen, die ein Histopathologe/in auch nutzen würde, um eine Klassifizierung zu machen. Die blauen, ovalen Strukturen sind Zellkerne, welche sich in malignen (=bösartigen) Zellen stark verändert darstellen (s. nächstes Kapitel)
Die Architektur des CNN bleibt gleich und es wird jedem Patch das Label des gesamten Schnittes zugewiesen. Also wenn der gesamte Schnitt vom Typ “Clear Cell” (CC) ist, bekommt jedes der Patches auch dieses Label.
Die Resultate sehen schonmal sehr vielversprechend aus:
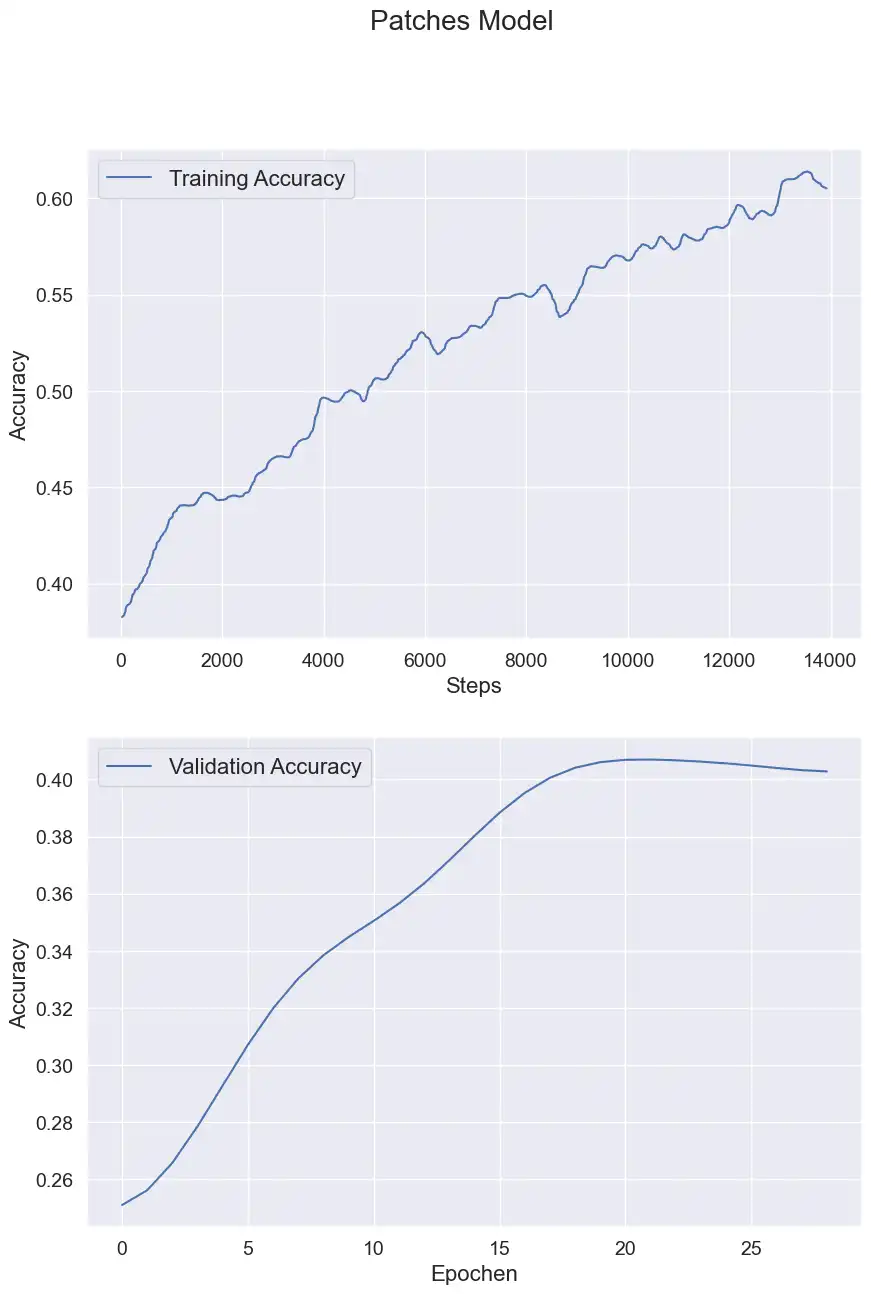
Hiermit wurde eine Validation Accuracy von über 40% erreicht. Wie genau sich die Performance in den einzelnen Klassen entwickelt kann man in dieser Animation verfolgen:
Hierbei ist Class 0 = CC, Class 1 = EC, Class 2 = HGSC, Class 3 = LGSC, Class 4 = MC. Man erkennt, wie das neuronale Netz immer weitere Features lernt, um die richtige Klassifizierung zu machen.
Nun kommen wir aber zu einer weiteren Herausforderung: Bei der UBC Challenge wurden die geheimen Testdaten, auf denen der finale Score berechnet wird, mit bewusst großer Abweichung ausgewählt. D.h. diese Testdaten wurden in anderen Laboratorien mit möglicherweise etwas anderen Methoden aufgenommen. Das neuronale Netz muss also sehr gut generalisieren können und trotz Änderungen in Farbe oder Größe die richtige Klassifizierung machen. Daher erreicht das Patches Model auf den Testdaten nur einen Score von 22%.
Bisher habe ich nur meine eigene CNN Architektur genutzt, die relativ einfach aufgebaut ist. Es gibt aber schon unzählige Architekturen im Computer Vision Bereich, die sehr gut performen, vor allem was Generalisierung anbelangt.
ResNet, ConvNet, EfficientNet… Was die großen Modelle können
Ein Klassiker unter den Computer Vision Models ist wohl das ResNet. In Klassifizierungen wie dem ImageNet erreicht diese Architektur bis heute sehr gute Ergebnisse. Doch an der Spitze sind aktuell die Vision Transformer Models wie EVA, Caformer, usw.
Es ist fast immer sinnvoll, eines der bewährten Modelle mit vortrainierten Parametern als Ausgangspunkt zu nutzen (Transfer Learning). Damit kann das neuronale Netz schon von Beginn an viele verschiedene Formen und Strukturen erkennen. Eine großartige Library dafür ist TIMM - damit hat man Zugriff auf über 1000 Modelle mit vortrainierten Parametern.
Ausgehend von den Patch Daten konnte ich diese Performance auf den Testdaten erreichen:
|
Architektur |
Balanced Accuracy (Test) |
|
ResNet34d |
0,33 |
|
TF EfficientNet B4 |
0,30 |
|
EdgeNext Base |
0,5 |
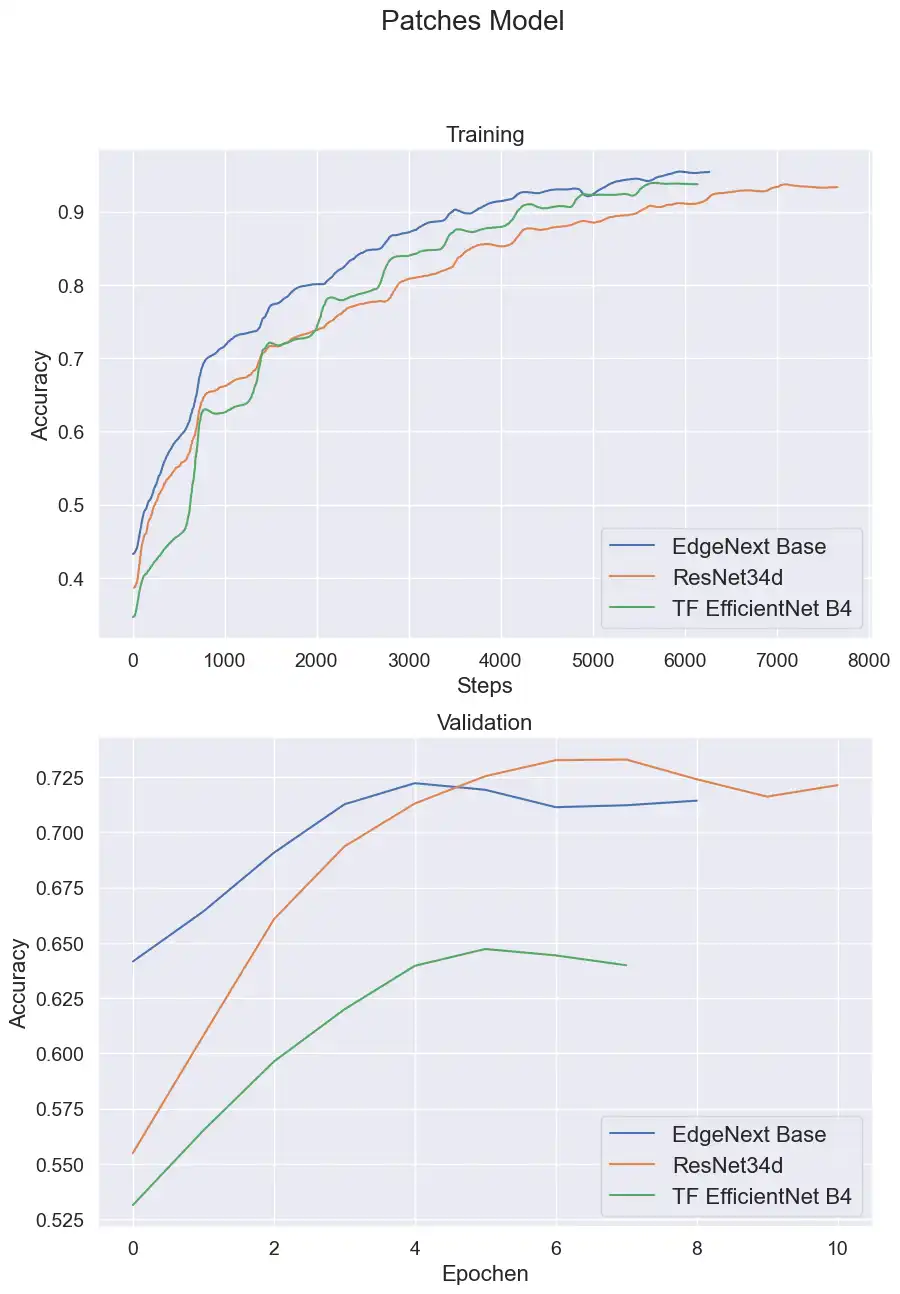
Das ist direkt mal eine Verdopplung der Performance! Das zeigt, wie wichtig der Startpunkt bei neuronalen Netzen ist. Ein untrainiertes Netz muss erst über viele Iterationen die richtigen Features in den Bildern finden, wohingegen die trainierten Netze schon direkt Formen und Farben erkennen und Lösungen finden können, die sonst gar nicht möglich wären.
Mit den 50% des EdgeNext bin ich zu dem Zeitpunkt der Submission Position 30 von über 1000 Teilnehmern, also unter den Top 3%!
Theoretisch ist das EdgeNext im Vergleich der anderen Modelle nicht die allerbeste Wahl. Wie oben schon beschrieben sind die Transformer Modelle noch besser, doch da gibt es ein Problem: Die Größe. Das EdgeNext hat 18 Millionen Parameter - klingt nach viel, aber die größten Transformer Modelle haben 1 Milliarde (!) Parameter. Und da ich jetzt nicht gerade eine RTX 6000 mit 48GB bei mir verbaut habe, musste ich den besten Kompromiss aus Leistung und Speicherbedarf wählen. Und genau dafür ist das EdgeNext auch gemacht: State-of-the-Art Performance bei minimalen Ressourcen. In meinen Tests hat sich das auch immer bestätigt.
Mit dem jetzigen Setup sind die Möglichkeiten meiner Meinung nach ziemlich ausgereizt. Um noch weiter voranzukommen, muss man besser verstehen, wie die Präparate überhaupt hergestellt werden und wie genau man die Krebsarten unterscheiden kann.
Histologie als Werkzeug in der Krebsforschung
Als Medizinphysiker ist die Histologie nicht mein Fachgebiet, daher hat das hier keinen Anspruch auf Vollständigkeit. Aber trotzdem sind Mikroskope ein durch und durch physikalisches Instrument. Daher möchte ich kurz das Vorgehen beschreiben, wie man zu den Bildern kommt, die wir oben schon gesehen haben (wer sich das genauer anschauen möchte, hier ein gutes Video dazu).
- Es wird eine Biopsie des betroffenen Gewebes entnommen. Je nach Organ kann das ein mehr oder weniger großer Eingriff sein. Außerdem entscheidet auch das Stadium des Tumors, also z.B. bei den hier relevanten Ovarialkarzinomen, ob nur Teile der Ovarien entfernt oder eine Hysterektomie durchgeführt wird. (Quelle)
- Im Labor werden die Gewebeproben fixiert, d.h. mit Formaldehyd durchtränkt, damit keine Zersetzungsprozesse das Gewebe zerstören.
- Die Prozessierung entzieht der Probe sämtliches Wasser und danach wird es in Paraffin-Wachs eingehüllt.
- Die Probe wird beim Slicing in sehr dünne (~1 𝛍m) Scheiben geschnitten, was durch das gehärtete Wachs erst möglich gemacht wird. Diese Scheiben werden auf einen Objektträger aus Glas aufgebracht.
- Das Präparat wird beim Staining eingefärbt, sodass Zellstrukturen besser zu sehen sind. Für biologische Präparate wird oft H&E verwendet. Hematoxylin und Eosin färben Proteine pink und den Zellkerne blau.
- Das fertige Präparat geht dann an Histopathologen/-innen zur Bewertung und wird dabei ggf. digitalisiert. Hierbei werden Lichtmikroskope genutzt, die das gefärbte Gewebe von unten beleuchten und dessen Objektive an der Oberseite für verschiedene Vergrößerungen eingestellt werden können.
Neben der Vergrößerung des Mikroskops gibt es weitere Einstellmöglichkeiten, z.B. ob man im Hell- oder Dunkelfeld arbeitet, ob ein Phasenkontrast eingesetzt wird usw. Das führt hier an der Stelle aber zu weit. Das Vorgehen beim Mikroskopieren startet immer bei einer geringen Vergrößerung, um sich einen Überblick zu verschaffen. Hat man dann einen relevanten Bereich erkannt, kann man dort die Vergrößerung erhöhen, um mehr Details zu erkennen.
Worauf muss man nun achten, wenn man die Schnittbilder mit Krebszellen betrachtet? Gleich vorweg: Zur sicheren Klassifizierung der Krebsarten braucht es erfahrene Mediziner, die sich in diesem Bereich über Jahre spezialisiert haben. Das kann man jetzt in einem kleinen Blogbeitrag nicht mal schnell in 5 min rüber bringen.
Was man allerdings mit etwas Übung gut erkennen kann, sind die Zellkern-Abnormalitäten. Diese Abweichungen treten in Krebszellen auf, da diese die Eigenschaft haben, sich unkontrolliert zu teilen. Dafür bedarf es sehr viel Proteinbiosynthese, die nur mit einem aktiven Zellkern möglich ist.
Ein normaler Zellkern…
- Hat eine regelmäßige Form
- Ist relativ klein (Zelle zu Kern Volumen Relation ungefähr 10)
- Hat eine glatte Oberfläche
- Hat kein oder kaum erkennbarer Nucleolus
Ein maligner Zellkern…
- Hat oft unregelmäßige und asymmetrische Formen
- Ist deutlich vergrößert, nimmt im Grenzfall sogar einen Großteil der Zelle ein
- Hat Einkerbungen und Unregelmäßigkeiten in der Oberfläche
- Hat ein deutlich erkennbaren Nucleolus
Am einfachsten lässt sich das an einem Beispiel erkennen:
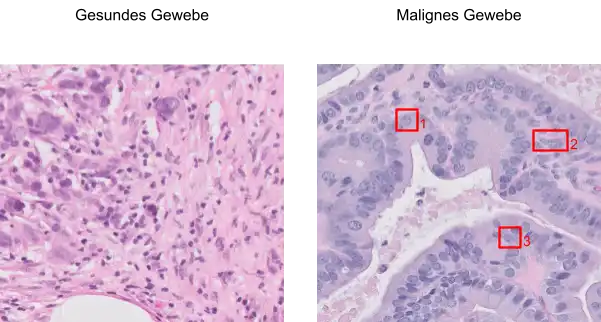
- Box 1: Die Zellkerne sind im Vergleich viel größer und nehmen einen Großteil der Zellen ein. Das gilt für alle Kerne im gesamten Bild.
- Box 2: Die Zellkerne sind z. T. unregelmäßig geformt.
- Box 3: Hier muss man etwas genauer hinzoomen. Der Nucleolus (dunkelblauer Punkt) ist deutlich zu erkennen.
Aus diesen Infos kann man sich jetzt zwei grundlegende Punkte für das weitere Vorgehen ableiten:
- Der Algorithmus sollte nur tatsächlich krebsartige Stellen für die Klassifizierung auswerten.
- Diese krebsartigen Stellen sollten so weit vergrößert sein, dass Zellkerne und ihre Form deutlich zu erkennen sind.
Krebszellen vs. Gesunde Zellen (Segmentierung)
Zu dem ersten Punkt: Eine Sache, die mir bei meiner Methode der Patch Extraktion von Anfang an zu Denken gegeben hat: Was ist, wenn das Präparat aus malignen und normalen Gewebe besteht? Tatsächlich ist es doch sogar eher unwahrscheinlich, dass eine ganze Probe nur aus Krebszellen besteht. Dann wären meine Patches entweder zufällig malignes Gewebe oder eben nicht. Und beim Training bekommt das neuronale Netz dann Bilder von komplett gesundem Gewebe und muss es einer Krebsart zuordnen. Das schafft selbst der beste Histopathologe der Welt nicht.
Circa ein Monat nach dem Start der Challenge haben die Leiter der Challenge zusätzlich Segmentierungs-Daten für einige der Schnittbilder veröffentlicht. Das sind Masken, die einem Teil der Pixel eine Klasse zuordnen. In diesem Fall malignes, gesundes oder abgestorbenes Gewebe.
Das hat es ermöglicht eine neue Methode für die Patch Extraktion zu nutzen:
- Das Originalbild wird in N x M Patches der Größe 512px aufgeteilt. Das wird hier ohne Abstand gemacht, d.h. die Patches liegen direkt nebeneinander.
- Aus der Maske wird der Gewebetyp ausgelesen und der Patch in ein entsprechenden Ordner einsortiert
Das Vorgehen wird klar wenn man sich das folgende Beispiel anschaut:

2 Stage Model: Erst Detektieren, dann Klassifizieren
Nachdem wir nun eindeutige Informationen darüber haben, welche Patches gesundes und welche malignes Gewebe zeigen, ist mein Ansatz ein 2 Stage Model. Also zwei Stages die nacheinander folgende Klassifizierungen vornehmen:
- Detektor: Ein neuronales Netz detektiert Patches, die malignes Gewebe zeigen. Dies ist dann eine binäre Klassifizierung aller Patches.
- Classifier: Ein zweites neuronales Netz bestimmt von allen als maligne detektierten Patches den Subtyp (HGSC, MC, …)
Nummer 1 hat natürlich die einfachere Aufgabe, doch die Schwierigkeit von weiter oben sollte hiermit gelöst sein: Der Classifier sieht nur noch tatsächlich malignes Gewebe und nicht wie zuvor auch gesunde Patches.
Für beide Stages wird wieder das EdgeNext benutzt, da es beim vorherigen Ansatz schon sehr gute Ergebnisse geliefert hat.
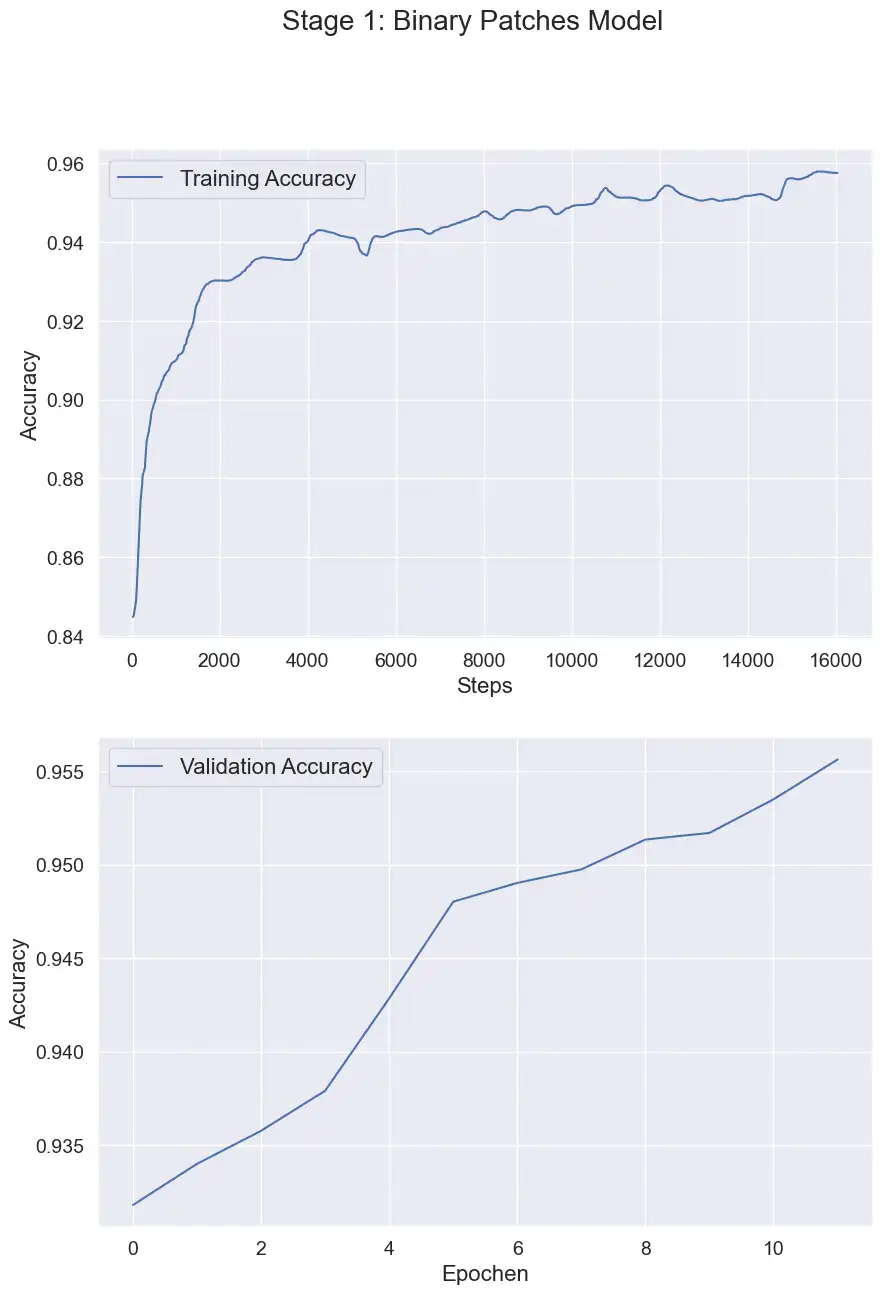
Wie zu erwarten, schafft es das neuronale Netz sehr gut, gesundes und malignes Gewebe zu unterscheiden. Jetzt bleibt nur noch die Frage ob Stage 2 auch die Unterscheidung in die Subtypen gut hinbekommt:
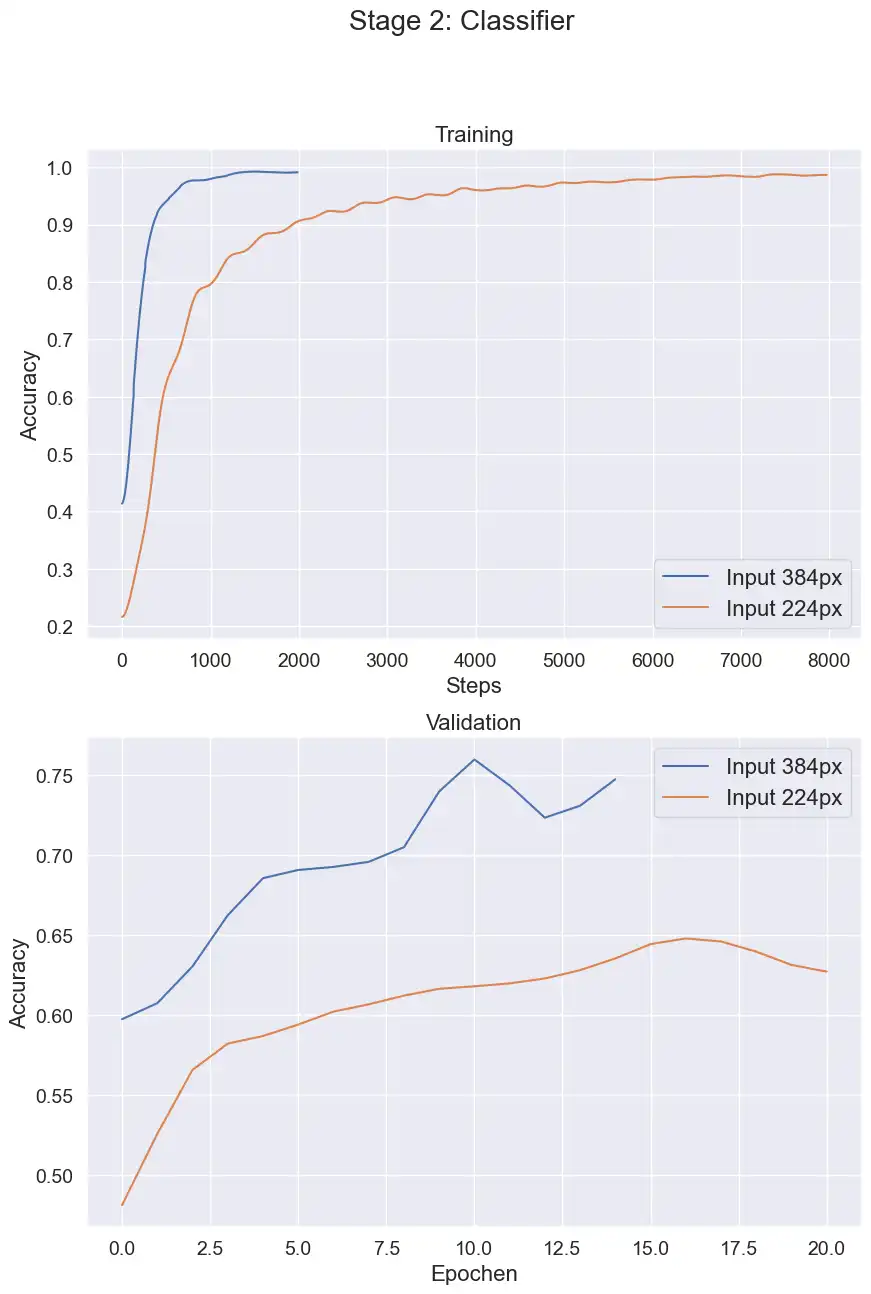
Bei diesem Training habe ich mehrere Auflösungen getestet. Das ursprüngliche EdgeNext wurde auf 320px x 320px großen Bildern trainiert. Die besten Ergebnisse mit den Patch Daten wurden mit 384px erzielt. Bei 224px gehen zu viele Details verloren und ein 512px Model zeigt keine Verbesserung.
Im Validierungs-Datensatz wurden ca. 75% erreicht. Interessanterweise ist das nur minimal besser als das erste Patches Model (~72%). Für das Testing wurde dann folgendes Vorgehen genutzt:
- Aus jedem Schnittbild werden M x N Patches mit der Größe 512px extrahiert.
- Komplett dunkle oder helle Patches werden aussortiert
- Detektion: Das Detection Model bestimmt für alle relevanten Patches, ob es sich um krebsartiges Gewebe handelt.
- Klassifizierung: Das Classifier Model nutzt alle krebsartigen Patches und bestimmt die Subtypen.
- Das Resultat ist eine Liste von Vorhersagen aller malignen Patches. Von dieser wird der Mittelwert bestimmt und als Endresultat in die Output-Tabelle geschrieben
Leider kommt das so implementierte Model nicht an das ursprüngliche Patches Model ran: 47% ist die Accuracy auf den Testdaten und damit liegt es mit 3% knapp dahinter.
Auch dieser Ansatz ist nicht perfekt - die Patches werden jetzt zuverlässig in malignes und gesundes Gewebe unterschieden und danach mit relativ guter Accuracy klassifiziert. Doch was ist mit Schritt 5? Angenommen, es gibt 10 krebsartige Patches, dann heißt das nicht, dass auf allen 10 auch der Subtyp (gut) zu erkennen ist.
Es müsste eine Möglichkeit geben, einem Machine Learning (ML) Ansatz alle relevanten Patches auf einmal zu "zeigen", damit aus mehreren Datenpunkten das Resultat berechnet werden kann…
Krebs KI als Multi Instance Learning: HGBC/RNN/t-SNE
Der große Vorteil von Convolutional Neural Networks ist, dass die Filter selbst erlernt werden. Schaut man sich die ResNet Architektur an, werden dort die Input Bilder in jedem Schritt runterskaliert und die Anzahl der Filter erhöht. Damit lernt das Netz Linien, Kreise und viele weitere komplexe Muster zu erkennen. Zum Schluss werden diese Werte in einen eindimensionalen Feature Vektor geschrieben und mit einem simplen Multi-Layer Perceptron klassifiziert.
So viel zur grauen Theorie, aber was soll uns das bei den Schnittbildern helfen? Oft werden die neuronalen Netze als Feature Extraktoren genutzt. D.h. jedes Bild ergibt einen kompakten Feature Vektor, der alle relevanten Informationen enthält. Damit kann man dann andere ML Algorithmen trainieren, wie z.B. ein rekurrentes neuronales Netz (RNN) oder einen Boosting Algorithmus.
Außerdem: Ein Feature Vektor ist viel einfacher zu handeln als ein Bild mit 512x512x3 Pixeln. Eine gute Methode, um die Daten zu visualisieren, nennt sich t-distributed stochastic neighbor embedding. Der etwas umständliche Name sagt im Grunde aus, dass die Datenpunkte so transformiert (embedded) werden, dass sie entsprechend ihrer Klasse maximal weit entfernt liegen. Die Dimension kann man dabei frei wählen, also auch 2D Daten, die einfach zu visualisieren sind. Lassen sich die Datenpunkte dann in dieser Darstellung gut unterscheiden, kann man davon ausgehen, dass ein ML Algorithmus das auch gut kann.
Das Vorgehen ist hier:
- Erstelle von jedem malignen Patch den Feature Vektor, basierend auf dem trainierten EdgeNext. Jeder Feature Vektor hat eine Dimension von 584.
- Von allen Feature Vektoren eines Schnittbildes wird das Maximum in jeder Feature Dimension berechnet. Ein Datenpunkt entspricht dann einem gesamten Schnittbild.
- Alle Daten werden mittels t-SNE dargestellt.
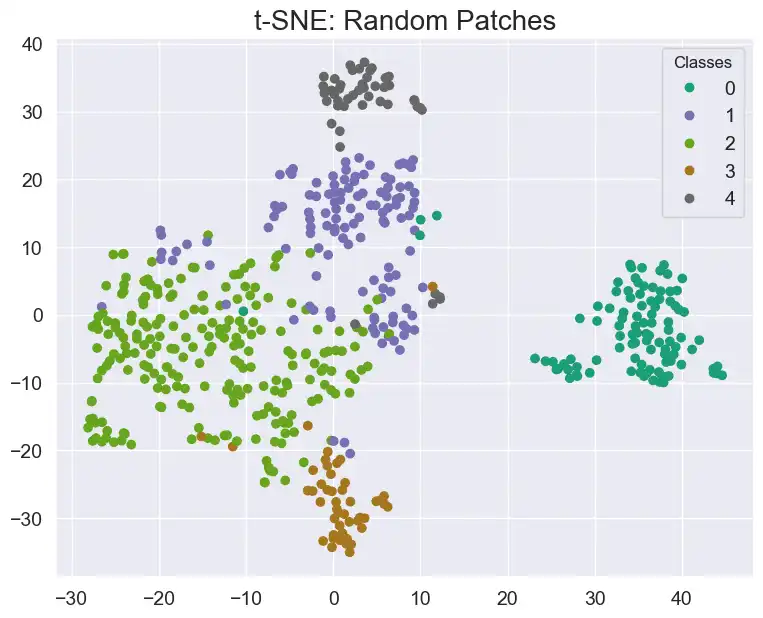
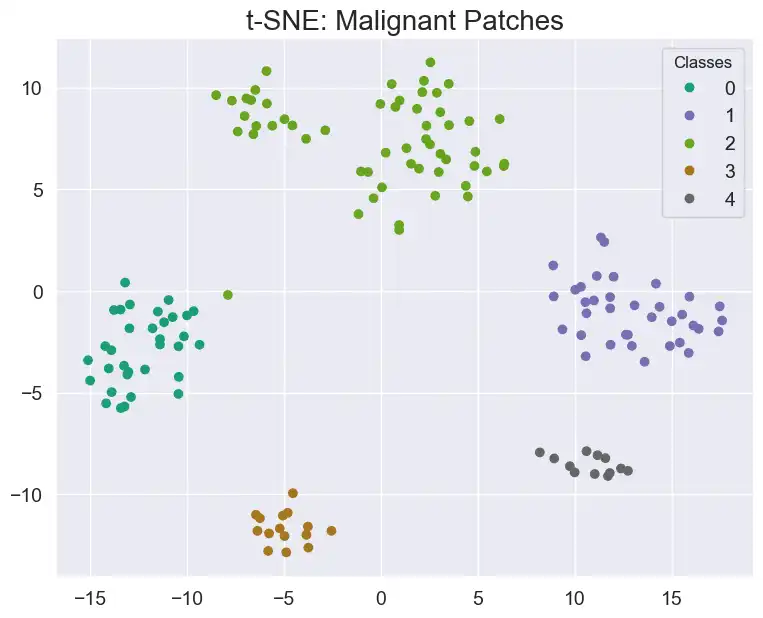
Man erkennt deutlich, dass die Datenpunkte bei den zufälligen Patches viel schwerer zu unterscheiden sind. Werden nur die malignen Patches genutzt, ist es einfach in der 2D Grafik die Grenzen zu finden. Das unterstützt die These, dass es keinen Sinn macht einfach zufällig extrahierte Patches zu nutzen, sondern die Masken mit einzubeziehen.
Basierend auf den malignen Patches können verschiedene Algorithmen trainiert werden:
|
Algorithmus |
Validation Accuracy |
|
Histogram Based Gradient Boosting (HGBC) |
100% |
|
K-Nearest Neighbor Classifier |
98% |
|
Support Vector Machine |
100% |
Die Ergebnisse sehen natürlich extrem gut aus, doch man muss bedenken, dass es nur ca. 150 Datenpunkte sind. Die Fähigkeit zur Generalisierung ist schwierig aus so wenig Daten rauszuholen.
Auf den Testdaten ergibt sich aber immerhin ein Score von 46% für den HGBC, was sehr nah an den neuronalen Netzen liegt.
Wrap it up: Was KI bei der Erkennung von Krebs tun kann
Bevor ich zum Fazit komme, erscheint mir das die richtige Stelle für ein kurzes Statement: Man hört immer wieder sehr viel Negatives über KI, zusammen mit irgendwelchen unsinnigen Katastrophengeschichten, die passieren könnten. Es gibt definitiv Gefahren und es muss klare Regelungen geben! Aber solange man sich in diesen Regelungen bewegt, ist KI ein Werkzeug, das uns in so vielen Lebensbereichen weiterhelfen kann. Sich dagegen komplett zu verschließen, kann definitiv nicht die Lösung sein.
Zum Ende dieser Challenge am 4. Januar 2024 habe ich hier mit dem zweiten Ansatz eines neuronalen Netzes eine Balanced Accuracy auf den Testdaten von 50% erreicht. Das entspricht einer Platzierung in den Top 5% !
Nachdem die Challenge abgeschlossen war, wurden die privaten Test Scores berechnet, also eine Auswertung mit bisher komplett ungenutzten Daten aus dem Testdatensatz. Da man nur zwei Submissions auswählen kann, die dann nach der Deadline für den privaten Score genutzt werden, hat sich die finale Platzierung etwas verschlechtert. Mit 44% bin ich aber immer noch in den Top 8% und habe damit auch eine Bronzemedaille.
Diese Challenge zeigt, wie viel Potential in den KI Methoden steckt. Die Aufgabe der Bestimmung von Subtypen ist sehr komplex und erfordert viel Erfahrung. Mit einem robusten Algorithmus können diese Krebsarten früher erkannt und vor allem auch früher behandelt werden.
Es gibt zwei Herausforderungen, die zum Schluss noch offen waren: Die Generalisierung und Outlier Detection.
Zur Generalisierung wäre es sinnvoll gewesen, weitere Datenquellen zu nutzen, um mehr Bilder und mehr Varianz zu bekommen. Da wir hier aber in einem sehr speziellen Feld sind, ist das nicht ohne Weiteres machbar. Auch die Segmentierung ist für Nicht-Histopathologen eine große Herausforderung.
Eine externe Datenquelle die ich getestset habe, war der Bevacizumab Response Datensatz. Leider hat sich dadurch keine Verbesserung in den Scores ergeben.
Der Standard für bessere Generalisierung ist Image Augmentation, also Variation in Helligkeit, Farbe, Kontrast, etc. der Bilder. Das wurde bei all meinen Experimenten angewendet, doch scheinbar reicht das nicht aus, um die Varianz in realen Daten abzubilden.
Außerdem war auffällig, dass der Unterschied von Training zu Validation zu Testing ziemlich groß war. Z.B. nahezu 100% im Training, 70% in der Validierung und nur noch 50% bei den Testdaten. Da würde man nicht unbedingt von Overfitting sprechen, da es in der Validierung nicht schlechter wurde, aber trotzdem muss es einen Grund geben.
Möglicherweise wurden Features gelernt, die in den Trainings-, und z. T. auch Validierungsdaten vorhanden waren, dafür aber viel weniger in den Testdaten. Ganz nach dem Prinzip von Occam’s Razor könnte es sein, dass einfach eine bestimmte Verteilung von roten und blauen Bereichen besonders oft bei einem Subtyp vorkommt. Das wäre sehr stark von dem Staining Prozess abhängig und nicht relevant für eine sinnvolle Klassifizierung.
Und zum zweiten Punkt: Der Outlier Detection. Für diesen Teil der Competition war zum Schluss nur noch wenig Zeit übrig. Ich habe eine One-Class SVM zur Novelty Detection getestet, was aber keine Verbesserung des Scores brachte.
Für beide Herausforderungen gilt wie so oft im ML Bereich: Mehr Daten! Eine andere Möglichkeit wäre ein größeres neuronales Netz gewesen, was ich, wie oben beschrieben, aufgrund der Hardwareanforderungen nicht sinnvoll nutzen konnte.
Ein paar Takeaways aus dieser Challenge:
- Benutze vortrainierte Netze: Die Menge an Informationen, die schon in diesen Models steckt, ist riesig, und man sollte definitiv davon Gebrauch machen.
- Nicht an Micro Optimierung aufhängen: Man tendiert gerne dazu, sehr viele minimale Änderungen (Learning Rate, Augmentation Parameter, …) auszuprobieren. Der Effekt ist meistens viel zu gering, als dass sich der Aufwand (und Zeit) lohnt.
- Gute Datengrundlage schaffen: Bevor man überhaupt mit dem KI-Teil startet, sollte man wenn möglich noch weitere Datenquellen hinzufügen und standardisieren.
Danke fürs Lesen und bei Fragen, kann man mich gerne per Mail oder Kontaktformular kontaktieren.